Avanços na compreensão da estrutura de patógenos
Estudo utiliza criomicroscopia eletrônica para examinar em detalhe proteínas de duas bactérias
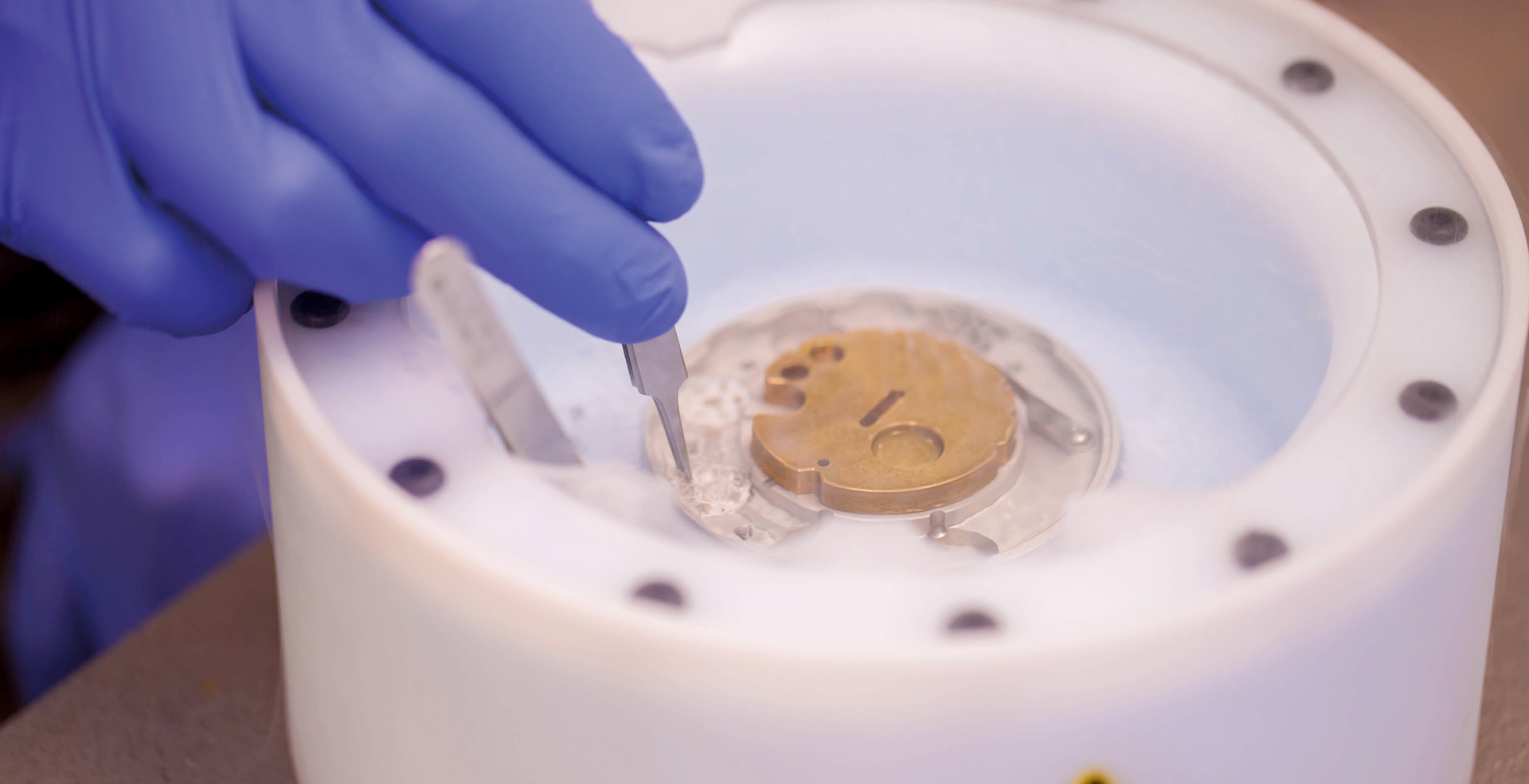
Uma atividade biológica natural, as trocas de DNA realizadas entre bactérias se tornaram um problema global. Devido ao mau uso de antibióticos, a habilidade bacteriana de absorver genes de outros organismos se tornou um veículo eficaz para o surgimento de superbactérias, que desafiam a medicina e os cientistas. Em sua pesquisa de pós-doutorado, Leonardo Talachia Rosa, professor do Instituto de Biologia (IB) da Unicamp, investigou como os mecanismos de resistência antimicrobiana se desenvolvem durante processos infecciosos. Seu trabalho, publicado recentemente na revista científica The EMBO Journal, detalha a estrutura molecular de proteínas envolvidas nesse processo e seu funcionamento, revelando sua importância para potencializar a aquisição de genes – e, assim, de resistência.
Rosa desenvolveu um estudo de biologia estrutural partindo da exploração da estrutura molecular, para entender um mecanismo biológico. A maior parte da pesquisa transcorreu no laboratório francês do Instituto Europeu de Química e Biologia (IECB, na sigla em francês), com a colaboração do bioquímico Rémi Fronzes – coautor do artigo recém-publicado. A decisão de adotar a criomicroscopia eletrônica (Cryo-EM, na sigla em inglês) como metodologia mostrou-se determinante para que o pesquisador decidisse realizar seu trabalho na França, uma vez que, à época, essa tecnologia ainda se encontrava em seus estágios iniciais no Brasil. Ao saber que a equipe liderada pelo cientista francês estava estudando estruturas de proteínas bacterianas usando a Cryo-EM, o brasileiro encontrou a oportunidade de unir o desejo de pesquisar o metabolismo de bactérias com o interesse em aprender uma técnica responsável por trazer resultados significativos.
Sua pesquisa se enquadra no campo da biologia estrutural e fornece novos detalhes sobre a atuação de proteínas em um processo denominado transformação natural bacteriana, durante o qual o patógeno realiza a captura e a incorporação de genes. “Quando estão colonizando um órgão, algumas bactérias são capazes de jogar toxinas no ambiente para matar as outras ao seu redor. Aquelas que morrem liberam seu DNA no entorno, e é esse material genético que pode ser parcialmente anexado ao genoma de quem as matou. Além de genes de resistência, nesses momentos, as bactérias captam novos fatores de virulência e genes que podem causar problemas no contexto hospitalar”, explica Rosa.
O biólogo trabalhou com proteínas presentes na Streptococcus pneumoniae e na Legionella pneumophila, ambas bactérias associadas a infecções do trato respiratório. A primeira provoca uma forma de pneumonia que figura, mundialmente, entre as principais causas de morte na primeira infância. Essa bactéria está associada, ainda, ao problema de infecções secundárias em hospitais. “Muitas vezes, a pessoa é internada por outro motivo, sara e depois morre de pneumonia”, afirma Rosa.
O segundo patógeno foi escolhido por pertencer a uma classe de bactérias evolutivamente distintas, as gram-negativas. “Essa é mais oportunista. De modo geral, não causa infecção em indivíduos, a não ser que estejam com seu sistema imunológico ao menos levemente comprometido”, esclarece.
Rosa focou moléculas de duas proteínas específicas: a RadA (presente na Streptococcus pneumoniae) e a ComM (encontrada na Legionella pneumophila). Ambas helicases, isto é, com participação ativa na transformação natural bacteriana. As duas atuam no interior da célula, separando a dupla fita de DNA da bactéria e criando um espaço para que os genes capturados externamente possam ser incorporados. “Essas duas proteínas aumentam a efetividade do processo em cem vezes. Se conseguirmos inibi-las, teremos uma redução de cem vezes em um mecanismo vinculado à aquisição de resistência antimicrobiana.”
Por trabalhar com a técnica Cryo-EM, o biólogo conseguiu examinar a estrutura molecular das helicases detalhadamente e, mais tarde, produzir um modelo 3D de cada uma. Rosa buscou reproduzi-las no estado mais próximo ao nativo, ou seja, na conformação assumida pelas proteínas no momento de suas atividades biológicas.
“Com o entendimento sobre como se localizam no espaço e de que forma desempenham sua função biológica, é possível construir desenhos mais inteligentes para novas drogas, que se ligarão a locais específicos no interior da proteína para tentar inibi-la ou alterar sua atuação, ajudando a anular os mecanismos que desempenham”, esclarece. Por ampliar o conhecimento sobre a evolução bacteriana, o trabalho indica caminhos possíveis para o desenvolvimento, no futuro próximo, de estratégias na criação de novos fármacos.
Semelhanças inesperadas
De volta à Unicamp, o biólogo se concentrou na interpretação dos modelos tridimensionais das duas proteínas. Para tanto, serviram de base conceitos teóricos da genética e da microbiologia. Embora as proteínas pesquisadas sejam diferentes e façam parte de bactérias diferentes (e que pertencem a classes distintas), Rosa curiosamente encontrou semelhanças em suas estruturas e formas de funcionar. Um dos principais achados deu-se na identificação, em ambas helicases, de uma parte praticamente igual de suas estruturas. Um pedaço normalmente não encontrado em outras proteínas que desempenham funções semelhantes. “Parece haver um indício de que essa parte extra, existente especificamente em proteínas dedicadas à transformação natural, ao processo de aquisição de genes, desempenha um papel importante”, afirma o pesquisador. Também supreenderam Rosa as semelhanças na relação entre o consumo energético das proteínas e o desempenho de suas funções biológicas – mais especificamente, no momento de aumentarem o espaço no qual a troca de informação genética acontece nas bactérias. O biólogo encontrou, ainda, locais de ligação para moléculas que fornecem energia e pôde explicar como essa energia é convertida na atividade biológica observada. “Nós fizemos algumas mutações, alteramos alguns componentes dentro da proteína e vimos como aquilo mudava ou não a sua atividade biológica. Para a RadA, identificamos um aminoácido que impactava muito significativamente a sua atuação. Quando esse aminoácido era alterado, a proteína queimava energia da célula, mas não conseguia realizar sua função biológica. Então, a eficiência da transformação natural caiu quase cem vezes, como se a proteína fosse apagada por completo”, descreve.
Potencial da criomicroscopia
O interesse do professor em usar a criomicroscopia eletrônica relaciona-se com o potencial da técnica, disseminada em países da Europa e nos Estados Unidos na década passada e, segundo Rosa, desde então responsável por avanços expressivos no campo da microbiologia. Sobretudo no que diz respeito ao conhecimento de estruturas moleculares de microrganismos. No Brasil, há três equipamentos aptos para a técnica, dos quais dois estão instalados no Centro Nacional de Pesquisa em Energia e Materiais (CNPEM), na Unicamp.
“Para se ter uma noção do grau de importância da Cryo-EM, os principais cientistas que contribuíram para seu desenvolvimento receberam o Prêmio Nobel de Química em 2017. Antes, praticamente não tínhamos novas descobertas por microscopia eletrônica, no nosso campo. Mas já no início da década passada, com a entrada da tecnologia, houve um boom. A quantidade de estruturas proteicas depositadas a cada ano passou a ser impressionante”, relata. A qualidade de resolução das imagens produzidas pelo microscópio, afirma o pesquisador, representa outro diferencial do equipamento. “O grau de detalhe do que se consegue ver ao fim do processo chega à escala de 1 angstrom [10-10 m] a 2 angstrom. É possível visualizar diferenças entre átomos dentro da estrutura proteica, o que vem permitindo a elucidação de estruturas biológicas cada vez mais complexas, com um maior grau de detalhamento.”
O primeiro passo para conseguir construir um modelo 3D das moléculas de RadA e ComM deu-se na captura de seus registros por diferentes ângulos e em diferentes conformações. O cientista usou o mesmo processo nos dois casos. Após isolar uma proteína de cada tipo em uma solução, Rosa congelou as amostras em nitrogênio líquido. O pesquisador explica que, dada a velocidade do processo de congelamento, as moléculas de água encontradas na solução não conseguem se remanejar a tempo de formarem cristais de gelo, resultando em um gelo cristalino e transparente, que permite visualizar com clareza moléculas praticamente em estado nativo.
Já para visualizar as estruturas moleculares, o biólogo utilizou um microscópio eletrônico especial. “Esse é um equipamento extremamente robusto, que trabalha com temperaturas também de nitrogênio líquido e possui uma diferença de potencial de 300 mil volts”, descreve. Ao passarem pelas amostras vitrificadas, os elétrons do microscópio registram a imagem daquilo que está congelado. Assim, obtêm-se exemplares da proteína em diferentes posições e ângulos. “Acabamos com milhões de imagens da mesma estrutura proteica em conformações variadas.”
Em um trabalho quase artesanal, o professor sobrepôs imagens da proteína para construir uma versão bidimensional. Após um tratamento estatístico, elaborou-se um mapa tridimensional, que permitiu ver como cada molécula se comporta. Com isso, Rosa conseguiu obter um modelo 3D das proteínas em um estado bem próximo do nativo – um fator fundamental para se compreender como as bactérias incorporam genes adquiridos externamente. “Na hora de desenvolver um fármaco para tratar uma infecção por bactéria, se o modelo usado não for o da proteína em seu estado nativo, o medicamento não vai se ligar à molécula biológica de forma adequada e não vai agir como deveria”, justifica.
Em Campinas, os dois aparelhos dedicados à criomicroscopia eletrônica já estão em operação, no Laboratório Nacional de Nanotecnologia (LNNano) do CNPEM. Utilizando o aparelho, pesquisadores da Unicamp e da Universidade de São Paulo (USP) desvendaram uma dezena de estruturas proteicas, conta Rosa. Atualmente, o professor se dedica a um novo projeto de biologia estrutural, no Laboratório de Bioquímica de Complexos Bacterianos (Labicobac) do IB, investigando o processo de infecção da bactéria causadora da coqueluche, uma doença reemergente no mundo.
Pesquisador associado do Centro de Pesquisa em Biologia de Bactérias e Bacteriófagos (Cepid B3) e jovem pesquisador da Fundação de Amparo à Pesquisa do Estado de São Paulo (Fapesp), o biólogo tem atuado, também, para promover a democratização do acesso ao Cryo-EM. “Estamos trabalhando para treinar o maior número possível de estudantes, a fim de que a técnica seja utilizada em toda a sua capacidade e a fim de que novas portas se abram nos estudos que temos desenvolvido dentro da Unicamp e do Estado de São Paulo.”
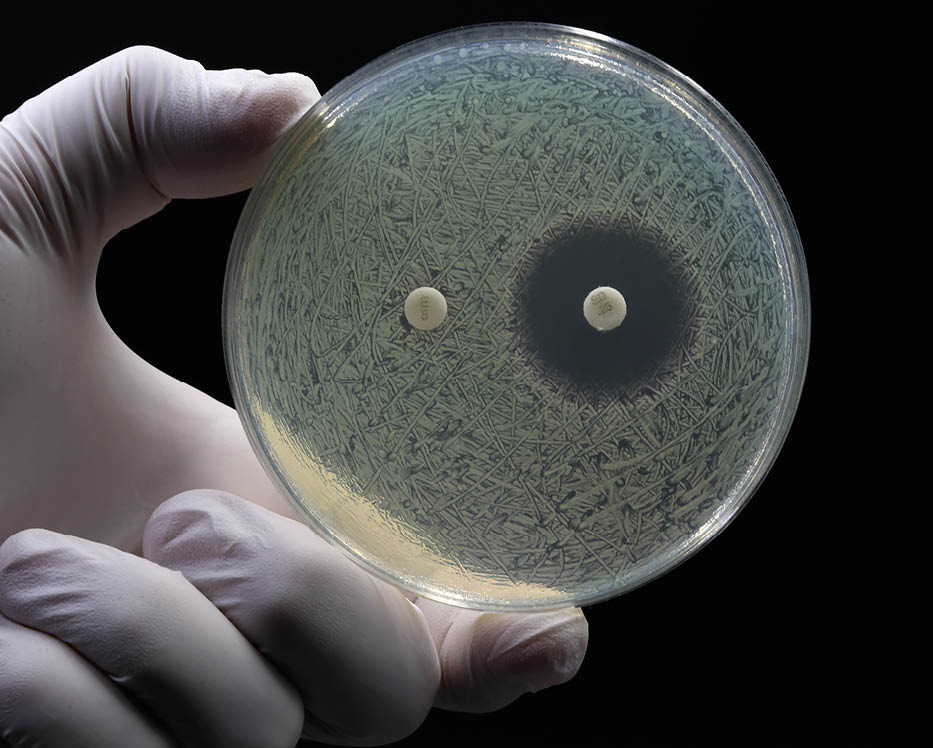
Desafio atual
Bactérias multirresistentes são a principal causa da sepse – infecção generalizada – nos hospitais brasileiros, de acordo com o Ministério da Saúde. A cada ano, registram-se 400 mil casos da doença entre adultos, dos quais mais da metade (60%) morre. Entre crianças, a taxa de mortalidade chega a 19%. O problema atinge o mundo todo. Segundo o último boletim de resistência aos antimicrobianos da Agência Nacional de Vigilância Sanitária (Anvisa), publicado neste ano com dados da Organização Mundial de Saúde (OMS), infecções causadas por superbactérias mataram mais de 1,2 milhão de pessoas em 2019. Das vítimas, um quinto tinha entre 0 e 5 anos de idade. Embora a situação resulte da ação humana, animais e vegetais também sofrem, pois são igualmente suscetíveis a doenças infecciosas.
Há vários fatores responsáveis por tornar um patógeno resistente a medicamentos que foram fabricados justamente para exterminá-lo. Tomar antibiótico de maneira indiscriminada e abandonar o tratamento antes do prazo representam comportamentos sabidamente relacionados com o aumento da capacidade de defesa da bactéria. O descarte incorreto de remédios e o uso de antibióticos na produção de alimentos também constam entre as práticas que contribuem para o fortalecimento dos patógenos.
Se mal administrados, esses remédios podem levar à seleção de bactérias resistentes à ação desses mesmos fármacos. Mesmo que já não estejam causando doenças dentro do corpo, de acordo com o biólogo, essas cepas funcionam como um reservatório de genes de resistência, pois processos como a transformação natural bacteriana torna possível incorporar materiais genéticos de novas bactérias. Assim, quando um desses patógenos entra no organismo de uma pessoa já infectada por outra bactéria, podem ocorrer novas trocas, fortalecendo ainda mais sua defesa contra drogas antimicrobianas.
O pesquisador vê na taxa de descobertas de novos antibióticos, aquém da demanda global, e na ineficácia de medicamentos lançados há mais de duas décadas outros aspectos que contribuem para a gravidade da situação. “Cada vez mais rápido, vão aparecendo novos tipos de resistência. O intervalo entre o aparecimento de um novo antibiótico e o surgimento de uma bactéria resistente a ele costumava ser de dez anos. Porém vi algumas pesquisas recentes dizendo que um ano após o lançamento de remédios já havia genes de resistência aparecendo.”
Encontrar soluções mais eficazes para enfrentar as superbactérias é um dos principais desafios da medicina e da ciência na atualidade. “Acredito que, juntamente com o combate ao câncer, as bactérias resistentes representam um dos maiores desafios da medicina atualmente. Isso é algo que já estamos enfrentando. Todo mundo conhece alguém que teve uma infecção e precisou recorrer a mais de uma medicação para tratá-la. É muito comum [nos hospitais] o médico receitar um antibiótico para um paciente e a medicação não funcionar, tendo, então, de usar dois ou três remédios. Ou fazer um antibiograma. Toda família tem pelo menos uma experiência parecida com essa para contar.”

Desafio atual
Bactérias multirresistentes são a principal causa da sepse – infecção generalizada – nos hospitais brasileiros, de acordo com o Ministério da Saúde. A cada ano, registram-se 400 mil casos da doença entre adultos, dos quais mais da metade (60%) morre. Entre crianças, a taxa de mortalidade chega a 19%. O problema atinge o mundo todo. Segundo o último boletim de resistência aos antimicrobianos da Agência Nacional de Vigilância Sanitária (Anvisa), publicado neste ano com dados da Organização Mundial de Saúde (OMS), infecções causadas por superbactérias mataram mais de 1,2 milhão de pessoas em 2019. Das vítimas, um quinto tinha entre 0 e 5 anos de idade. Embora a situação resulte da ação humana, animais e vegetais também sofrem, pois são igualmente suscetíveis a doenças infecciosas.
Há vários fatores responsáveis por tornar um patógeno resistente a medicamentos que foram fabricados justamente para exterminá-lo. Tomar antibiótico de maneira indiscriminada e abandonar o tratamento antes do prazo representam comportamentos sabidamente relacionados com o aumento da capacidade de defesa da bactéria. O descarte incorreto de remédios e o uso de antibióticos na produção de alimentos também constam entre as práticas que contribuem para o fortalecimento dos patógenos.
Se mal administrados, esses remédios podem levar à seleção de bactérias resistentes à ação desses mesmos fármacos. Mesmo que já não estejam causando doenças dentro do corpo, de acordo com o biólogo, essas cepas funcionam como um reservatório de genes de resistência, pois processos como a transformação natural bacteriana torna possível incorporar materiais genéticos de novas bactérias. Assim, quando um desses patógenos entra no organismo de uma pessoa já infectada por outra bactéria, podem ocorrer novas trocas, fortalecendo ainda mais sua defesa contra drogas antimicrobianas.
O pesquisador vê na taxa de descobertas de novos antibióticos, aquém da demanda global, e na ineficácia de medicamentos lançados há mais de duas décadas outros aspectos que contribuem para a gravidade da situação. “Cada vez mais rápido, vão aparecendo novos tipos de resistência. O intervalo entre o aparecimento de um novo antibiótico e o surgimento de uma bactéria resistente a ele costumava ser de dez anos. Porém vi algumas pesquisas recentes dizendo que um ano após o lançamento de remédios já havia genes de resistência aparecendo.”
Encontrar soluções mais eficazes para enfrentar as superbactérias é um dos principais desafios da medicina e da ciência na atualidade. “Acredito que, juntamente com o combate ao câncer, as bactérias resistentes representam um dos maiores desafios da medicina atualmente. Isso é algo que já estamos enfrentando. Todo mundo conhece alguém que teve uma infecção e precisou recorrer a mais de uma medicação para tratá-la. É muito comum [nos hospitais] o médico receitar um antibiótico para um paciente e a medicação não funcionar, tendo, então, de usar dois ou três remédios. Ou fazer um antibiograma. Toda família tem pelo menos uma experiência parecida com essa para contar.”






